-
Die Universität
- Herzlich willkommen
- Das sind wir
- Medien & PR
-
Studium
- Allgemein
- Studienangebot
- Campusleben
-
Forschung
- Profil
- Infrastruktur
- Kooperationen
- Services
-
Karriere
- Arbeitgeberin Med Uni Graz
- Potenziale
- Arbeitsumfeld
- Offene Stellen
-
Diagnostik
- Patient*innen
- Zuweiser*innen
-
Gesundheitsthemen
- Gesundheitsinfrastruktur
Forschungsteam Pritišanac
Forschungsschwerpunkt: Entzündungen, Autoimmunität und Krebs
Etwa 60% der menschlichen Proteine enthalten lange Abschnitte ohne stabile Sekundär- oder Tertiärstrukturen, die als intrinsisch ungeordnete Proteinregionen (IDRs) bekannt sind. Diese Regionen werden zunehmend als Schlüsselregulatoren für ein ganzes Spektrum zellulärer Prozesse anerkannt, darunter mRNA-Transkription, Spleißen, Proteintranslation, Signaltransduktion und Lokalisierung in spezifischen biomolekularen Kondensaten. Darüber hinaus sind IDRs in Risikogenen angereichert, die mit komplexen Krankheiten in Verbindung gebracht werden, einschließlich neurologischer Entwicklungsstörungen und Krebs (Tsang, Pritišanac et al. 2020 Cell). Um ihre Funktionen auszuführen, gehen IDRs intra- und intermolekulare Wechselwirkungen ein, die in der Regel transient und hochdynamisch sind und können ihre Grenzflächen je nach Bindungspartner anpassen (Alderson & Pritišanac, et al. 2023 PNAS).
Unsere Forschung zielt darauf ab, das Verständnis der molekularen Erkennung in den dynamischen Bereichen des Proteoms und Transkriptoms zu vertiefen. Durch die Kombination von computergestützter Strukturbiologie, modernsten KI-Modellen, Bioinformatik und Protein-NMR-Spektroskopie versuchen wir, abstimmbare biomolekulare Interaktionen zu verstehen, vorherzusagen und zu designen, die für Gesundheit und Krankheitspathologie entscheidend sind. Gemeinsam mit unseren Kooperationspartner*innen führen wir Forschungsarbeiten durch, die von der Grundlagenforschung über die Entdeckung von Arzneimitteln und die Interpretation von Krankheitsvarianten bis hin zum Protein-Engineering reichen.
Netzwerk: Neben der Zusammenarbeit innerhalb des Otto-Loewi-Forschungszentrums und der Medizinischen Universität Graz arbeiten wir mit Forscher*innen aus der ganzen Welt zusammen. Zu unseren Kooperationspartner*innen gehören Reid Alderson (Helmholtz Munich), Lu-Yang Wang (SickKids Toronto), Jason Moffat (University of Toronto), Anne Conibear (TU Wien), Vaclav Veverka (Charles University) und Matteo Degiacomi (Durham University).
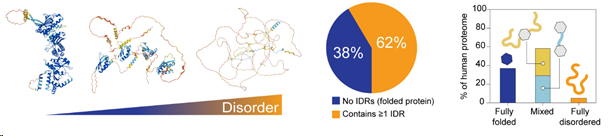
Einzelne Proteine können ein Spektrum an intrinsischer struktureller Unordnung aufweisen (links). Im menschlichen Proteom enthalten über 60% der Proteine mindestens eine intrinsisch ungeordnete Region (Mitte). Ungeordnete Regionen können entweder als Termini auftreten, die strukturierte Domänen flankieren, oder als Verbindungsglieder zwischen strukturierten Domänen (rechts). Ein kleiner Prozentsatz (~5%) des menschlichen Proteoms sind vollständig ungeordnete Proteine.


